CRISPR
| Cascade (CRISPR-associated complex for antiviral defense – Phức hệ đi kèm với CRISPR cho phòng thủ chống lại virus) | |
|---|---|
| |
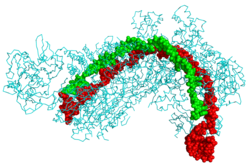
| Protein CRISPR Cascade (xanh dương) bám vào CRISPR RNA (xanh lục) và DNA của virus (đỏ)[1] | |
| Danh pháp | |
| Sinh vật | |
| Ký hiệu | CRISPR |
| PDB | 4QYZ |
| Dữ liệu khác | |
| Một phần của loạt bài về |
| Kỹ thuật di truyền |
|---|
 |
| Sinh vật biến đổi gen |
| Lịch sử và quy định |
| Quy trình |
| Ứng dụng |
| Tranh cãi |
CRISPR (/ˈkrɪspər/; tiếng Anh: clustered regularly interspaced short palindromic repeats) là một họ các trình tự DNA được tìm thấy trong bộ gen của các sinh vật nhân sơ như vi khuẩn và vi khuẩn cổ.[2] Những trình tự này hình thành từ các đoạn DNA của những thể thực khuẩn từng tấn công vào sinh vật nhân sơ đó. Chúng được dùng để phát hiện và phá hủy DNA của những loại thực khuẩn tương tự trong các lần tấn công về sau. Do đó, chúng đóng vai trò quan trọng trong hệ thống phòng thủ virus (phage) của sinh vật nhân sơ.[2]

Hệ CRISPR-Cas là một hệ miễn dịch ở sinh vật nhân sơ mang lại khả năng chống lại các yếu tố di truyền ngoại lai, như sự xâm nhập của plasmid và thể thực khuẩn,[4][5][6] tạo thành một dạng miễn dịch thu được. RNA bắt cặp với trình tự của vùng đệm (spacer) trên DNA mới xâm nhập và giúp protein Cas (CRISPR-associated) nhận diện và thực hiện cắt đứt sợi DNA xâm nhập gây bệnh. Những protein Cas khác do RNA dẫn đường cắt các RNA ngoại lai xâm nhập.[7] CRISPR được tìm thấy trong khoảng 50% các bộ gen vi khuẩn và gần 90% các bộ gene vi khuẩn cổ.[8]
Những hệ thống này đã dẫn đến kỹ thuật chỉnh sửa gen CRISPR sử dụng gen cas9.[9] Quá trình chỉnh sửa này có ứng dụng rộng rãi bao gồm nghiên cứu sinh học, phát triển các sản phẩm công nghệ sinh học, và chữa trị các bệnh.[10][11][12] Kỹ thuật chỉnh sửa gen CRISPR-Cas9 là nguyên nhân chính giúp Emmanuelle Charpentier và Jennifer Doudna nhận Giải Nobel Hóa học năm 2020.[13][14]
Lịch sử
[sửa | sửa mã nguồn]Chuỗi lặp lại
[sửa | sửa mã nguồn]Sự phát hiện các trình tự DNA lặp lại xảy ra độc lập ở ba nơi khác nhau trên thế giới. Năm 1987, nhà nghiên cứu Yoshizumi Ishino và đồng nghiệp từ Đại học Osaka lần đầu tiên mô tả thứ mà sau này được gọi CRISPR. Họ vô tình nhân đôi một đoạn CRISPR trong quá trình tái bản gen "iap".[15] Các đoạn lặp lại này được sắp xếp rất kỳ lạ. Những chuỗi lặp lại thường xếp liên tiếp nhau, không có những chuỗi khác xen giữa.[12][15] Họ không biết công dụng của những đoạn lặp thành cụm bị gián đoạn này.
Năm 1993, các nhà nghiên cứu tại Hà Lan đưa ra hai bài báo về một cụm các đoạn lặp trực tiếp (DR) bị gián đoạn trong vi khuẩn Mycobacterium tuberculosis. Họ nhận thấy các chuỗi xen giữa vào những đoạn lặp trực tiếp này khác nhau tùy vào chủng M. tuberculosis[16] và dùng tính chất này để phát triển phương pháp nhận dạng spoligotyping, vẫn được dùng đến ngày nay.[17][18]
Francisco Mojica tại Đại học Alicante ở Tây Ban Nha nghiên cứu các đoạn lặp lại và chức năng của chúng trong các loài vi khuẩn cổ Haloferax và Haloarcula. Người hướng dẫn của Mojica vào lúc đó cho rằng những đoạn lặp lại có vai trò trong việc phân chia các DNA đã nhân đôi vào hai tế bào con trong quá trình phân bào, bởi plasmid và nhiễm sắc thể với những đoạn lặp giống nhau không thể cùng tồn tại trong Haloferax volcanii. Việc phiên mã những đoạn lặp gián đoạn này cũng được nghiên cứu lần đầu tiên, dẫn đến việc những đặc trưng của CRISPR.[18][19] Năm 2000, Mojica khảo sát các nguồn khoa học và một trong những sinh viên tìm kiếm trong các bộ gen đã được công bố. Họ xác định được những đoạn lặp gián đoạn ở 20 loài vi sinh vật cùng thuộc một họ.[20] Năm 2001, Mojica và Ruud Jansen trong khi đang tìm kiếm những đoạn lặp gián đoạn khác, đề xuất cái tên CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats; tạm dịch: Đoạn lặp palindrome ngắn gián đoạn thường xuyên theo cụm) nhằm tránh nhầm lẫn xuất phát từ nhiều tên gọi khác được dùng trong giới khoa học để diễn tả những đoạn DNA này.[19][21] Năm 2002, Tang, et al. tìm ra bằng chứng cho thấy những vùng lặp CRISPR trong bộ gen của Archaeoglobus fulgidus được phiên mã thành phân tử RNA dài rồi được biến thành những đoạn RNA ngắn, cùng các chuỗi dài 2, 3 hoặc nhiều hơn đơn vị đệm.[22]
Phức hệ CRISPR
[sửa | sửa mã nguồn]Một bước tiến lớn trong việc hiểu rõ CRISPR đến từ quan sát của Jansen rằng những cụm lặp lại ở sinh vật nhân sơ này đi kèm với một tập hợp các gen tương đồng tạo thành các phức hệ CRISPR hay gen cas. Bốn gen cas (cas 1–4) được nhận diện từ đầu. Các protein Cas chứa motif cấu trúc helicase và nuclease, gợi ý về vai trò trong cấu trúc động của các locus CRISPR.[23] Cái tên CRISPR được dùng thống nhất cho hình mẫu này. Tuy nhiên, chức năng của chúng vẫn chưa được biết đến.

Năm 2005, ba nhóm nghiên cứu độc lập chỉ ra rằng một số đoạn đệm CRISPR xuất phát từ DNA thể thực khuẩn và DNA ngoại nhiễm sắc thể như là plasmid.[27][28][29] Nói cách khác, các đoạn đệm (spacer) là những đoạn DNA lấy từ những virus từng cố gắng xâm nhập tế bào trước đó. Nguồn gốc của các đoạn đệm là dấu hiệu cho thấy phức hệ CRISPR/cas có thể đóng một vai trò trong hệ miễn dịch thu được ở vi khuẩn.[24][30] Cả ba nghiên cứu đưa ra ý tưởng này ban đầu bị các tạp chí có danh tiếng từ chối, nhưng cũng xuất hiện trong những tạp chí khác.[31]
Nghiên cứu đầu tiên, bởi Mojica và đồng nghiệp tại Đại học Alicante,[28] tiên đoán vai trò của mã RNA của đoạn đệm trong việc nhận diện mục tiêu, một quá trình có thể tương tự với hệ thống can thiệp RNA trong tế bào nhân thực. Koonin và đồng nghiệp mở rộng giả thuyết can thiệp RNA này, đề xuất cơ chế hoạt động của các loại CRISPR-Cas khác nhau dựa trên chức năng được dự đoán của protein tạo ra từ chúng.[32]
Các thí nghiệm của nhiều nhóm khác nhau cho thấy cơ chế cơ bản của miễn dịch CRISPR-Cas. Năm 2007, bằng chứng thực nghiệm đầu tiên cho thấy CRISPR là một hệ miễn dịch thu được được đăng tải.[5][12] Một vùng CRISPR trong Streptococcus thermophilus lấy đoạn đệm từ DNA của một thể thực khuẩn xâm nhập. Các nhà nghiên cứu kiểm soát khả năng kháng các loại phage khác nhau của S. thermophilus bằng cách thêm và loại bỏ các đoạn đệm có trình tự giống với các phage đó.[33][34] Năm 2008, Brouns và Van der Oost tìm ra một phức hệ các protein Cas (gọi là Cascade) trong E. coli cắt tiền RNA CRISPR trong những đoạn lặp thành những phân tử RNA trưởng thành chứa đoạn đệm gọi là RNA CRISPR (crRNA), gắn với phức hệ protein.[35] Hơn nữa, người ta phát hiện rằng cần có Cascade, crRNA và một helicase/nuclease (Cas3) để giúp vi khuẩn miễn dịch với một virus DNA. Cùng năm đó, Marraffini và Sontheimer xác nhận rằng một trình tự CRISPR trong S. epidermidis nhắm vào DNA chứ không phải RNA để tránh giao nạp. Phát hiện này trái với những cơ chế tương tự can thiệp RNA được đề ra trước đó cho miễn dịch CRISPR-Cas, mặc dù một hệ thống CRISPR-Cas nhắm vào RNA ngoại lai được tìm thấy trong Pyrococcus furiosus.[12][33] Một nghiên cứu năm 2010 cho thấy CRISPR-Cas cắt cả sợi thể thực khuẩn và DNA plasmid trong S. thermophilus.[36]
Cas9
[sửa | sửa mã nguồn]Các nhà nghiên cứu tập trung vào một hệ CRISPR đơn giản trong Streptococcus pyogenes dựa vào protein Cas9. Endonuclease Cas9 là một phức hệ bốn phần gồm hai phân tử crRNA nhỏ và RNA CRISPR CRISPR RNA chuyển hoạt (tracrRNA).[37][38] Jennifer Doudna và Emmanuelle Charpentier thiết kế lại endonuclease Cas9 thành một hệ dễ kiểm soát hơn bằng cách hợp hai phân tử RNA lại thành một RNA dẫn đường, mà khi kết hợp với Cas9, có thể tìm và cắt DNA chỉ định bởi RNA dẫn đường. Đóng góp này có ý nghĩa quan trọng và được vinh danh bởi Giải Nobel Hóa học năm 2020. Bằng cách điều khiển chuỗi nucleotide của RNA dẫn đường, phức hệ Cas9 nhân tạo có thể được lập trình để đánh vào bất kỳ chuỗi DNA nào xâm nhập.[39] Một nhóm khác gồm Virginijus Šikšnys và đồng nghiệp cho thấy Cas9 từ hệ CRISPR của S. thermophilus có thể được tái lập trình để nhắm vào mục tiêu tùy ý bằng cách thay đổi trình tự của crRNA. Những bước tiến này thúc đẩy các nỗ lực chỉnh sửa bộ gen bằng hệ thống CRISPR-Cas9 tùy biến.[18]
Những nhóm dẫn đầu bởi Trương Phong và George Church cùng đăng tải mô tả việc chỉnh sửa gen trong tế bào người sử dụng CRISPR-Cas9 lần đầu tiên.[12][40][41] Từ đó kỹ thuật này đã được dùng cho nhiều loại sinh vật, bao gồm nấm men (Saccharomyces cerevisiae),[42][43][44] mầm bệnh cơ hội Candida albicans,[45][46] cá ngựa vằn (Danio rerio),[47] ruồi giấm thường (Drosophila melanogaster),[48][49] kiến (Harpegnathos saltator[50] và Ooceraea biroi[51]), muỗi (Aedes aegypti[52]), giun tròn (Caenorhabditis elegans),[53] thực vật,[54] chuột,[55][56] khỉ[57] và phôi người.[58]
CRISPR đã được chỉnh sửa để làm yếu tố phiên mã lập trình được, cho phép các nhà khoa học tập trung và kích hoạt hoặc vô hoạt các gen nhất định.[59]
Cas12a (trước là Cpf1)
[sửa | sửa mã nguồn]Năm 2015, nuclease Cas12a (trước gọi là Cpf1[60]) được miêu tả trong phức hệ CRISPR/Cpf1 của vi khuẩn Francisella novicida.[61][62] Tên gọi gốc của nó, từ một họ protein TIGRFAM định nghĩa năm 2012, cho thấy sự phổ biến loại CRISPR-Cas này ở chi Prevotella và Francisella. Cas12a khác với Cas9 ở một số điểm bao gồm: gây ra vết cắt 'so le' trong đoạn DNA kép thay vì vết cắt 'thẳng' như của Cas9, dựa trên một trình tự PAM giàu thymine và chỉ cần một RNA CRISPR (crRNA) để tìm thấy mục tiêu, trong khi Cas9 cần cả crRNA và một crRNA chuyển hoạt (tracrRNA).
Sự khác nhau này có thể khiến Cas12a tốt hơn Cas9 ở một số mặt. Ví dụ, các crRNA nhỏ của Cas12a phù hợp cho chỉnh sửa gen phức tạp, do số lượng crRNA sử dụng được lớn hơn snRNA của Cas9. Ngoài ra, đầu 5′ của Cas12a có thể được dùng cho việc lắp ghép DNA với độ chính xác cao hơn so với việc tái bản enzyme hạn chế thường dùng.[63] Cuối cùng, Cas12a chẻ đôi DNA cách điểm PAM xuôi dòng 18–23 cặp base, nghĩa là không gây xáo trộn trình tự nhận diện sau khi chỉnh sửa, cho phép chẻ DNA nhiều lần. Ngược lại, Cas9 cắt 3 cặp base ngược dòng điểm PAM, dẫn đến đột biến thêm bớt làm phá hủy trình tự nhận diện, ngăn không cho cắt tiếp nữa. Về lý thuyết, những lần cắt DNA liên tiếp có thể tăng khả năng cho việc chỉnh sửa gen diễn ra như mong muốn.[64]
Cas13 (trước là C2c2)
[sửa | sửa mã nguồn]Năm 2016, nuclease Cas13a (trước gọi là C2c2) ở vi khuẩn Leptotrichia shahii được miêu tả. Cas13 là một endonuclease RNA do RNA dẫn đường, nghĩa là nó không nhắm vào DNA mà chỉ cắt RNA. Cas13 được dẫn đường bởi crRNA của nó tới mục tiêu ssRNA và bám vào rồi cắt mục tiêu. Một tính chất đặc trưng của Cas13 so với Cas9 là sau khi cắt, Cas13 vẫn bám vào mục tiêu rồi cắt những phân tử ssRNA khác.[65] Tính chất này được gọi là "collateral cleavage" và đã được sử dụng để phát triển một số công nghệ chẩn đoán.[66][67][68]
Sử dụng cho chỉnh sửa gene
[sửa | sửa mã nguồn]Một phiên bản đơn giản của hệ CRISPR/Cas, gọi là CRISPR/Cas9, đã được áp dụng làm kỹ thuật chỉnh sửa bộ gene. Bằng cách đưa vào trong một tế bào phức hệ nuclease Cas9 với một RNA dẫn đường (gRNA) tổng hợp, có thể cắt bộ gene của tế bào tại những vị trí mong muốn, cho phép loại bỏ những gene hiện có và/hoặc thêm vào những đoạn DNA mới.[69][70][71] Phức hệ Cas9-gRNA tương ứng với phức CAS III CRISPR-RNA trong hình vẽ minh họa bên cạnh.

Kỹ thuật chỉnh sửa gene CRISPR/Cas có nhiều ứng dụng tiềm năng, bao gồm trong y học và nâng cao năng suất cây trồng trong nông nghiệp. Áp dụng phức hệ CRISPR/Cas9-gRNA cho chỉnh sửa bộ gene[72][73] đã được ban biên tập của tạp chí Science thuộc AAAS lựa chọn là đột phá khoa học của năm 2015.[74] Đã nổi lên những đề cập về đạo đức trong sinh học liên quan đến sử dụng khả năng của CRISPR cho chỉnh sửa dòng mầm (germline) ở động vật và người.[75]
Cấu trúc locus
[sửa | sửa mã nguồn]Đoạn lặp và đoạn đệm
[sửa | sửa mã nguồn]Một vị trí (locus) CRISPR được cấu tạo từ một trình tự dẫn đầu giàu AT, theo sau là những đoạn lặp (repeat) ngắn ngăn cách bởi các đoạn đệm (spacer) khác nhau.[76] Đoạn lặp CRISPR thường có kích cỡ từ 28 đến 37 cặp base (bp), tuy nhiên có trường hợp chúng ít đến 23 bp và nhiều đến 55 bp.[77] Một số có đối xứng dyad, dấu hiệu của cấu trúc bậc hai như thân-vòng ('kẹp tóc') thường thấy trong RNA, còn những loại khác thì không có cấu trúc cụ thể. Kích cỡ của các đoạn đệm trong những đoạn CRISPR khác nhau thường là 32–38 bp, dao động trong khoảng 21 đến 72 bp.[77] Những đoạn đệm mới có thể xuất hiện nhanh do phản ứng miễn dịch với virus xâm nhập.[78] Thông thường một đoạn CRISPR có ít hơn 50 đơn vị trình tự lặp-đệm.[77]
Tổng quan
[sửa | sửa mã nguồn]CRISPR/Cas9 – những chiếc kéo phân tử làm từ enzyme và RNA
[sửa | sửa mã nguồn]Những chiếc kéo cắt gene, các dao mổ phân tử – những thuật ngữ mô tả này được sử dụng để dành cho việc truyền tải nội dung của phương pháp mới về chỉnh sửa hơn là cái tên CRISPR-Cas9 khá khó mà có thể làm được.[79] Như tên gọi đề xuất, hệ thống, trong đó, ở dạng tự nhiên của nó, bao gồm hai phân tử RNA và một phân tử protein, có thể tách phân tử di truyền DNA. Hơn nữa, nó có thể làm điều này với độ chính xác phẫu thuật tại một vị trí cụ thể trong bộ gen. Điều này cho phép các nhà nghiên cứu làm tắt biểu hiện gene hoặc chèn các trình tự mới tại vị trí cắt. Kết quả là, DNA có thể được sửa đổi nhanh hơn và dễ dàng hơn nhiều so với khả năng sử dụng các phương pháp chỉnh sửa gene trước đó.[74][79][80]
Mặc dù hệ thống về cơ bản có vẻ đơn giản, nhưng các yếu tố khác nhau phải được phối hợp với độ chính xác cực cao để kéo cắt gene có thể hoạt động với độ chính xác như vậy. Vì lý do này, ngay cả sau 30 năm nghiên cứu, chức năng của CRISPR-Cas9 vẫn chưa được hiểu hoàn toàn.[79][80]
Trình tự xuôi ngược trong bộ gene
[sửa | sửa mã nguồn]3'-... C T T A A G...-5'
Một trình tự trong sợi xoắn kép DNA mà đọc xuôi ở sợi này giống hệt với đọc ngược ở sợi bổ sung kia. Tại đây, enzyme EcoR1 sẽ nhận ra và thực hiện cắt ở trình tự này.
Câu tiếng Anh "Able was I ere I saw Elba" là một câu xuôi ngược (palindrome), nghĩa là khi đọc ngược lại từ phải sang trái được nội dung giống hệ giống như khi đọc theo chiều thuận. Sự khởi đầu của cuộc cách mạng CRISPR đã được đánh dấu bằng việc phát hiện ra một số lượng lớn các trình tự xuôi ngược lặp đi lặp lại trong một vùng DNA của vi khuẩn. Trong các trình tự này, các chữ cái của mã di truyền, bốn phân tử base adenine, cytosine, thymine và guanine, được sắp xếp sao cho chúng có thứ tự giống như ở sợi DNA bổ sung khi đọc theo hướng ngược lại.[79] Cũng vì đặc điểm này mà tên gọi CRISPR được viết tắt từ những chữ cái đầu của cụm từ khá dài Clustered Regularly Interspaced Short Palindromic Repeats, tạm dịch: Cụm các trình tự xuôi ngược ngắn có độ dài bằng nhau lặp lại đan xen giữa các vùng đệm.[74][81]
Không giống như từ xuôi ngược, ví dụ ‘civic’ và ‘tenet’, mà có ý nghĩa, các đoạn xuôi ngược trong từ điển di truyền không được sử dụng để dịch mã thành các protein chức năng. Mặc dù vậy, chúng không phải hoàn toàn là không có ý nghĩa.[79] Các enzyme cắt DNA (nuclease) thường tìm đến các trình tự xuôi ngược này như là các trình tự nhận diện, và tại đây chúng thực hiện cắt phân tử DNA. Các trình tự này thường dài khoảng 4, 6 hoặc 8 base, mặc dù một số protein cắt đòi hỏi đến 20 cặp base hoặc nhiều hơn.[74][81]
Bên cạnh các trình tự xuôi ngược ngắn lặp lại giống nhau của CRISPR là các vùng mang thông tin phiên mã cho phân tử RNA, có sự sắp xếp rất ổn định (cấu trúc bậc hai). Chúng dài khoảng 23 đến 47 cặp base. Có thể tìm thấy các vùng có độ dài biến đổi này nằm xen lẫn các trình tự xuôi ngược.[79] Những đoạn mang thông tin phiên mã này có nguồn gốc từ bộ gene của DNA ngoại lai xâm nhập vào tế bào vi khuẩn, và được gọi là vùng đệm DNA (spacer DNA).[74][81]
Vùng CRISPR còn bao gồm một vùng khởi động (promoter) đảm bảo cho vùng CRISPR được đọc và phiên mã thành CRISPR-RNA (crRNA). Ngoài ra còn có các gene đi kèm với CRISPR (CRISPR-associated genes, Cas) nằm bên cạnh vùng khởi động.[79] Những gene mã hóa cho việc tổng hợp lên các protein Cas - hay chính là các enzyme có chức năng cắt sợi DNA. Các trình tự CRISPR và vùng đệm theo sau bởi một vùng phiên mã thành phân tử RNA gọi là tracrRNA, mà nó hướng dẫn các phân tử cắt và crRNA đến vị trí đích trên phân tử DNA của virus.[74][81]
Chức năng tự nhiên của CRISPR-Cas
[sửa | sửa mã nguồn]Chúng ta - loài người - thường coi vi khuẩn là một trong những tác nhân gây bệnh. Nhưng vi khuẩn cũng gặp phải tình trạng ốm yếu. Một số virus thực sự chuyên tấn công những vi sinh vật này. Có thể coi những virus đó, mà được biết đến là thể thực khuẩn, như hình ảnh giống với những tàu vũ trụ đổ bộ xuống một hành tinh ở xa.[79] Virus tiêm DNA của chúng vào tế bào vi khuẩn, mà sau đó được tái bản, và sinh ra thể thực khuẩn mới - quá trình này có thể giết chết tế bào vật chủ. Giống như các sinh vật khác, vi khuẩn cũng đã phát triển các cơ chế khéo léo nhằm bảo vệ chúng khỏi những kẻ xâm lược này. Các hệ thống CRISPR-Cas là một trong những cơ chế như vậy.[74][82]
Phòng ngừa miễn dịch với danh sách ghi nhớ
[sửa | sửa mã nguồn]Các tế bào vi khuẩn có thể sử dụng phức hệ CRISPR-Cas để bảo vệ chúng khỏi những lần xâm lăng về sau, vì CRISPR-Cas tạo cho hệ thống phòng thủ tiêm nhiễm của vi khuẩn một loại danh sách ghi nhớ: khi một thể thực khuẩn bám vào màng tế bào vi khuẩn và sau đó tiêm DNA của nó vào bên trong tế bào, phức hệ phòng thủ sẽ sao chép một đoạn ngắn những DNA ngoại lai này và gắn vào những đoạn trình tự CRISPR đã có trước đó trên DNA của vi khuẩn.[79] Các vùng đệm (spacer) - có trình tự với độ dài khác nhau nằm giữa các đoạn CRISPR có độ dài bằng nhau - do vậy tạo nên một kiểu danh sách thư viện lưu trữ mọi chủng loại tác nhân gây bệnh mà tế bào vi khuẩn từng gặp phải.[74][82] Chỉ 1 vi khuẩn trong 10 triệu vi khuẩn bị virus tấn công có khả năng thu nạp vùng đệm để tăng khả năng bảo vệ của hệ miễn dịch của nó.[79]
Vi khuẩn cũng truyền lại bộ thư viện này cho thế hệ con cháu của chúng. Một số nhà nghiên cứu coi điều này như là sự xác nhận giả thuyết phát triển bởi nhà sinh học Jean-Baptiste de Lamarck trong thế kỷ XIX, mà hầu như bị phản đối bởi đa số các nhà khoa học trong thời gian dài. Theo học thuyết của ông, các đặc tính thu nạp được trong vòng đời của sinh vật có thể được truyền sang cho các thế hệ sau.[74][82]
Một nửa số vi khuẩn đã biết ngày nay và hầu như mọi loài vi khuẩn cổ đều có hệ thống phòng thủ CRISPR-Cas. Cách tế bào triển khai hệ thống này phụ thuộc vào từng loài vi sinh vật. Về cơ bản có hai lớp CRISPR-Cas đã được các nhà sinh học biết đến và có thể chia nhỏ hai lớp này thành những lớp con. Hệ thống Lớp 1 chứa các phức hệ protein bao gồm nhiều phân tử, trong khi hệ thống Lớp 2 chỉ có một protein có chức năng cắt DNA.[74][82]
Nhưng trên tất cả chúng đều có đặc điểm chung đó là chúng chèn thêm các đoạn DNA ngoại lai mới vào vùng CRISPR và phiên mã các phân tử RNA từ các vùng này, mà sau đó phân tử RNA này sẽ hướng dẫn một enzyme hoặc phức hệ enzyme đến đoạn DNA đích.[79] Các phân tử RNA có thể bám vào một vị trí trên DNA xâm nhập mà khớp bổ sung với chính trình tự của chúng và nhờ đó hướng dẫn cho enzyme Cas nơi nó tiến hành cắt DNA ngoại lai. Cơ chế này làm DNA từ virus trở lên vô hại và vi khuẩn tránh khỏi sự lây nhiễm.[74][82]
CRISPR-Cas9 là một trong những hệ chức năng có cấu hình đơn giản nhất và khiến cho nó rất phù hợp trong việc ứng dụng ở lĩnh vực công nghệ sinh học. Với CRISPR-Cas9 và tracrRNA, phức hệ chỉ cần hai phân tử RNA và một protein Cas9 để tìm đến DNA đích và cắt đứt đoạn này.[79] Các nhà sinh học thậm chí có thể tổng hợp hai phân tử RNA này thành một phân tử RNA dẫn đường duy nhất trong phòng thí nghiệm, do đó tạo thành một phức hệ sử dụng dễ dàng hơn nữa. Thêm vào đó, CRISPR-Cas9 hoạt động không chỉ ở vi khuẩn mà còn làm việc tốt trong các tế bào nhân thực. Từ giun kim cho đến con người - những chiếc kéo cắt gene mới khám phá này có thể sử dụng ở khắp mọi nơi.[74][82]
Sự hoạt động của CRISPR-Cas9
[sửa | sửa mã nguồn]Với hệ CRISPR-Cas9, các trình tự CRISPR và vùng đệm được phiên mã thành CRISPR-RNA (crRNA). Trước khi phân tử này có thể dẫn protein Cas9 đến vị trí cắt DNA, nó phải được chỉnh sửa thành dạng cuối cùng nhờ các enzyme cắt và một số phần của nó bị cắt bỏ. RNase III là một trong những enzyme như thế. Cùng với trình tự tracrRNA nó biến đổi dạng ban đầu của crRNA thành một phân tử đầy đủ chức năng.[74][83]
crRNA thành thục chứa một trình tự phiên mã của CRISPR và của DNA ngoại lai.[79] Trình tự này cung cấp cho Cas9 trình tự nhận diện mà tại vị trí đó trên DNA mới xâm nhập sẽ bị cắt. crRNA bám vào tracrRNA, và chỉ cần hai phân tử này có thể cho Cas9 biết vị trí nó thực hiện cắt phân tử DNA.[74][83]
Trình tự nhận diện khớp bổ sung với crRNA là chưa đủ để cho Cas9 có thể bám vào DNA sợi ngoại lai: nó cũng cần một "môtip nằm cạnh tiền vùng đệm" (‘proto-spacer adjacent motif’) hay viết tắt là PAM. Cas9 chỉ có thể bám vào sợi xoắn kép DNA nếu một trình tự PAM có ba nucleotide có chứa hai guanine và một base bất kỳ khác nằm cạnh trình tự nhận diện.[83]
Hai sợi của DNA bị tháo xoắn và phân tử crRNA/tracrRN – hoặc RNA dẫn đường nhân tạo – có thể gắn kèm vào.[79] Enzyme sau đó thực hiện cắt hai sợi DNA ở cùng một vị trí. Do vậy, để Cas9 có thể cắt DNA, cần phải có cả trình tự nhận diện và đoạn môtip PAM. Bởi vì bộ gene của vi khuẩn không có bất kỳ một đoạn PAM nào, nó được bảo vệ khỏi bị phá hủy bởi chính hệ miễn dịch của nó.[74][83]
Tham khảo
[sửa | sửa mã nguồn]- ^ Mulepati S, Héroux A, Bailey S (2014). “Crystal structure of a CRISPR RNA–guided surveillance complex bound to a ssDNA target”. Science. 345 (6203): 1479–1484. Bibcode:2014Sci...345.1479M. doi:10.1126/science.1256996. PMC 4427192. PMID 25123481.
- ^ a b Barrangou R (2015). “The roles of CRISPR-Cas systems in adaptive immunity and beyond”. Current Opinion in Immunology. 32: 36–41. doi:10.1016/j.coi.2014.12.008. PMID 25574773.
- ^ Horvath P, Barrangou R (tháng 1 năm 2010). “CRISPR/Cas, the immune system of bacteria and archaea”. Science. 327 (5962): 167–170. Bibcode:2010Sci...327..167H. doi:10.1126/science.1179555. PMID 20056882. S2CID 17960960.
- ^ Redman M, và đồng nghiệp (tháng 8 năm 2016). “What is CRISPR/Cas9?”. Archives of Disease in Childhood. Education and Practice Edition. 101 (4): 213–5. doi:10.1136/archdischild-2016-310459. PMC 4975809. PMID 27059283.
- ^ a b Barrangou, Rodolphe; Fremaux, Christophe; Deveau, Hélène; Richards, Melissa; Boyaval, Patrick; Moineau, Sylvain; Romero, Dennis A.; Horvath, Philippe (23 tháng 3 năm 2007). “CRISPR provides acquired resistance against viruses in prokaryotes”. Science (New York, N.Y.). 315 (5819): 1709–1712. doi:10.1126/science.1138140. ISSN 1095-9203. PMID 17379808.
- ^ Marraffini, Luciano A.; Sontheimer, Erik J. (19 tháng 12 năm 2008). “CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA”. Science (New York, N.Y.). 322 (5909): 1843–1845. doi:10.1126/science.1165771. ISSN 1095-9203. PMC 2695655. PMID 19095942.
- ^ Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J (2016). “Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems” (PDF). Science. 353 (6299): aad5147. doi:10.1126/science.aad5147. hdl:1721.1/113195. PMID 27493190. S2CID 11086282.
- ^ Hille F, và đồng nghiệp (tháng 3 năm 2018). “The Biology of CRISPR-Cas: Backward and Forward”. Cell. 172 (6): 1239–1259. doi:10.1016/j.cell.2017.11.032. PMID 29522745.
- ^ Zhang F, Wen Y, Guo X (2014). “CRISPR/Cas9 for genome editing: progress, implications and challenges”. Human Molecular Genetics. 23 (R1): R40–6. doi:10.1093/hmg/ddu125. PMID 24651067.
- ^ CRISPR-CAS9, TALENS and ZFNS - the battle in gene editing https://www.ptglab.com/news/blog/crispr-cas9-talens-and-zfns-the-battle-in-gene-editing/
- ^ Nakade, S; Yamamoto, T; Sakuma, T (ngày 4 tháng 5 năm 2017). “Cas9, Cpf1 and C2c1/2/3-What's next?”. Bioengineered. 8 (3): 265–273. doi:10.1080/21655979.2017.1282018. ISSN 2165-5979. PMC 5470521. PMID 28140746.
- ^ a b c d e Hsu PD, Lander ES, Zhang F (tháng 6 năm 2014). “Development and applications of CRISPR-Cas9 for genome engineering”. Cell. 157 (6): 1262–1278. doi:10.1016/j.cell.2014.05.010. PMC 4343198. PMID 24906146.
- ^ “Press release: The Nobel Prize in Chemistry 2020”. Nobel Foundation. Truy cập ngày 7 tháng 10 năm 2020.
- ^ Wu, Katherine J.; Peltier, Elian (ngày 7 tháng 10 năm 2020). “Nobel Prize in Chemistry Awarded to 2 Scientists for Work on Genome Editing – Emmanuelle Charpentier and Jennifer A. Doudna developed the Crispr tool, which can alter the DNA of animals, plants and microorganisms with high precision”. The New York Times. Truy cập ngày 7 tháng 10 năm 2020.
- ^ a b Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A (tháng 12 năm 1987). “Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product”. Journal of Bacteriology. 169 (12): 5429–5433. doi:10.1128/jb.169.12.5429-5433.1987. PMC 213968. PMID 3316184.
- ^ van Soolingen D, de Haas PE, Hermans PW, Groenen PM, van Embden JD (tháng 8 năm 1993). “Comparison of various repetitive DNA elements as genetic markers for strain differentiation and epidemiology of Mycobacterium tuberculosis”. Journal of Clinical Microbiology. 31 (8): 1987–1995. doi:10.1128/JCM.31.8.1987-1995.1993. PMC 265684. PMID 7690367.
- ^ Groenen PM, Bunschoten AE, van Soolingen D, van Embden JD (tháng 12 năm 1993). “Nature of DNA polymorphism in the direct repeat cluster of Mycobacterium tuberculosis; application for strain differentiation by a novel typing method”. Molecular Microbiology. 10 (5): 1057–1065. doi:10.1111/j.1365-2958.1993.tb00976.x. PMID 7934856.
- ^ a b c Mojica FJ, Montoliu L (2016). “On the Origin of CRISPR-Cas Technology: From Prokaryotes to Mammals”. Trends in Microbiology. 24 (10): 811–820. doi:10.1016/j.tim.2016.06.005. PMID 27401123.
- ^ a b Mojica FJ, Rodriguez-Valera F (2016). “The discovery of CRISPR in archaea and bacteria” (PDF). The FEBS Journal. 283 (17): 3162–3169. doi:10.1111/febs.13766. hdl:10045/57676. PMID 27234458.
- ^ Mojica FJ, Díez-Villaseñor C, Soria E, Juez G (tháng 4 năm 2000). “Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria”. Molecular Microbiology. 36 (1): 244–246. doi:10.1046/j.1365-2958.2000.01838.x. PMID 10760181.
- ^ Barrangou, Rodolphe; van der Oost, John (2013). CRISPR-Cas Systems: RNA-mediated Adaptive Immunity in Bacteria and Archaea. Heidelberg: Springer. tr. 6. ISBN 978-3-642-34656-9.
- ^ Charpentier E, Richter H, van der Oost J, White MF (tháng 5 năm 2015). “Biogenesis pathways of RNA guides in archaeal and bacterial CRISPR-Cas adaptive immunity”. FEMS Microbiology Reviews. 39 (3): 428–441. doi:10.1093/femsre/fuv023. PMC 5965381. PMID 25994611.
- ^ Jansen R, Embden JD, Gaastra W, Schouls LM (tháng 3 năm 2002). “Identification of genes that are associated with DNA repeats in prokaryotes”. Molecular Microbiology. 43 (6): 1565–1575. doi:10.1046/j.1365-2958.2002.02839.x. PMID 11952905.
- ^ a b c Horvath P, Barrangou R (tháng 1 năm 2010). “CRISPR/Cas, the immune system of bacteria and archaea”. Science. 327 (5962): 167–170. Bibcode:2010Sci...327..167H. doi:10.1126/Science.1179555. PMID 20056882. S2CID 17960960.
- ^ Marraffini LA, Sontheimer EJ (tháng 3 năm 2010). “CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea”. Nature Reviews Genetics. 11 (3): 181–190. doi:10.1038/nrg2749. PMC 2928866. PMID 20125085.
- ^ Grissa I, Vergnaud G, Pourcel C (tháng 5 năm 2007). “The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats”. BMC Bioinformatics. 8: 172. doi:10.1186/1471-2105-8-172. PMC 1892036. PMID 17521438.
- ^ Pourcel C, Salvignol G, Vergnaud G (tháng 3 năm 2005). “CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies”. Microbiology. 151 (Pt 3): 653–663. doi:10.1099/mic.0.27437-0. PMID 15758212.
- ^ a b Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (tháng 2 năm 2005). “Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements”. Journal of Molecular Evolution. 60 (2): 174–182. Bibcode:2005JMolE..60..174M. doi:10.1007/s00239-004-0046-3. PMID 15791728. S2CID 27481111.
- ^ Bolotin A, Quinquis B, Sorokin A, Ehrlich SD (tháng 8 năm 2005). “Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin”. Microbiology. 151 (Pt 8): 2551–2561. doi:10.1099/mic.0.28048-0. PMID 16079334.
- ^ Morange M (tháng 6 năm 2015). “What history tells us XXXVII. CRISPR-Cas: The discovery of an immune system in prokaryotes”. Journal of Biosciences. 40 (2): 221–223. doi:10.1007/s12038-015-9532-6. PMID 25963251.
- ^ Lander ES (tháng 1 năm 2016). “The Heroes of CRISPR”. Cell. 164 (1–2): 18–28. doi:10.1016/j.cell.2015.12.041. PMID 26771483.
- ^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (tháng 3 năm 2006). “A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action”. Biology Direct. 1: 7. doi:10.1186/1745-6150-1-7. PMC 1462988. PMID 16545108.
- ^ a b Marraffini LA (tháng 10 năm 2015). “CRISPR-Cas immunity in prokaryotes”. Nature. 526 (7571): 55–61. Bibcode:2015Natur.526...55M. doi:10.1038/nature15386. PMID 26432244. S2CID 3718361.
- ^ Pennisi E (tháng 8 năm 2013). “The CRISPR craze”. News Focus. Science. 341 (6148): 833–836. Bibcode:2013Sci...341..833P. doi:10.1126/science.341.6148.833. PMID 23970676.
- ^ Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, Dickman MJ, Makarova KS, Koonin EV, van der Oost J (tháng 8 năm 2008). “Small CRISPR RNAs guide antiviral defense in prokaryotes”. Science. 321 (5891): 960–964. Bibcode:2008Sci...321..960B. doi:10.1126/science.1159689. PMC 5898235. PMID 18703739.
- ^ Garneau JE, Dupuis MÈ, Villion M, Romero DA, Barrangou R, Boyaval P, và đồng nghiệp (tháng 11 năm 2010). “The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA”. Nature. 468 (7320): 67–71. Bibcode:2010Natur.468...67G. CiteSeerX 10.1.1.451.9645. doi:10.1038/nature09523. PMID 21048762. S2CID 205222849.
- ^ Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E (tháng 3 năm 2011). “CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III”. Nature. 471 (7340): 602–607. Bibcode:2011Natur.471..602D. doi:10.1038/nature09886. PMC 3070239. PMID 21455174.
- ^ Barrangou R (tháng 11 năm 2015). “Diversity of CRISPR-Cas immune systems and molecular machines”. Genome Biology. 16: 247. doi:10.1186/s13059-015-0816-9. PMC 4638107. PMID 26549499.
- ^ Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E (tháng 8 năm 2012). “A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity”. Science. 337 (6096): 816–821. Bibcode:2012Sci...337..816J. doi:10.1126/science.1225829. PMC 6286148. PMID 22745249.
- ^ Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F (tháng 2 năm 2013). “Multiplex genome engineering using CRISPR/Cas systems”. Science. 339 (6121): 819–823. Bibcode:2013Sci...339..819C. doi:10.1126/science.1231143. PMC 3795411. PMID 23287718.
- ^ Mali P, Yang L, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM (tháng 2 năm 2013). “RNA-guided human genome engineering via Cas9”. Science. 339 (6121): 823–826. Bibcode:2013Sci...339..823M. doi:10.1126/science.1232033. PMC 3712628. PMID 23287722.
- ^ DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM (tháng 4 năm 2013). “Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems”. Nucleic Acids Research. 41 (7): 4336–4343. doi:10.1093/nar/gkt135. PMC 3627607. PMID 23460208.
- ^ Zhang GC, Kong II, Kim H, Liu JJ, Cate JH, Jin YS (tháng 12 năm 2014). “Construction of a quadruple auxotrophic mutant of an industrial polyploid saccharomyces cerevisiae strain by using RNA-guided Cas9 nuclease”. Applied and Environmental Microbiology. 80 (24): 7694–7701. doi:10.1128/AEM.02310-14. PMC 4249234. PMID 25281382.
- ^ Liu JJ, Kong II, Zhang GC, Jayakody LN, Kim H, Xia PF, Kwak S, Sung BH, Sohn JH, Walukiewicz HE, Rao CV, Jin YS (tháng 4 năm 2016). “Metabolic Engineering of Probiotic Saccharomyces boulardii”. Applied and Environmental Microbiology. 82 (8): 2280–2287. doi:10.1128/AEM.00057-16. PMC 4959471. PMID 26850302.
- ^ Vyas VK, Barrasa MI, Fink GR (2015). “Candida albicans CRISPR system permits genetic engineering of essential genes and gene families”. Science Advances. 1 (3): e1500248. Bibcode:2015SciA....1E0248V. doi:10.1126/sciadv.1500248. PMC 4428347. PMID 25977940.
- ^ Ng H, Dean N (2017). “Candida albicans by Increased Single Guide RNA Expression”. mSphere. 2 (2): e00385–16. doi:10.1128/mSphere.00385-16. PMC 5397569. PMID 28435892.
- ^ Hwang WY, Fu Y, Reyon D, Maeder ML, Tsai SQ, Sander JD, Peterson RT, Yeh JR, Joung JK (tháng 3 năm 2013). “Efficient genome editing in zebrafish using a CRISPR-Cas system”. Nature Biotechnology. 31 (3): 227–229. doi:10.1038/nbt.2501. PMC 3686313. PMID 23360964.
- ^ Gratz SJ, Cummings AM, Nguyen JN, Hamm DC, Donohue LK, Harrison MM, Wildonger J, O'Connor-Giles KM (tháng 8 năm 2013). “Genome engineering of Drosophila with the CRISPR RNA-guided Cas9 nuclease”. Genetics. 194 (4): 1029–1035. doi:10.1534/genetics.113.152710. PMC 3730909. PMID 23709638.
- ^ Bassett AR, Tibbit C, Ponting CP, Liu JL (tháng 7 năm 2013). “Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system”. Cell Reports. 4 (1): 220–228. doi:10.1016/j.celrep.2013.06.020. PMC 3714591. PMID 23827738.
- ^ Yan H, Opachaloemphan C, Mancini G, Yang H, Gallitto M, Mlejnek J, Leibholz A, Haight K, Ghaninia M, Huo L, Perry M, Slone J, Zhou X, Traficante M, Penick CA, Dolezal K, Gokhale K, Stevens K, Fetter-Pruneda I, Bonasio R, Zwiebel LJ, Berger SL, Liebig J, Reinberg D, Desplan C (tháng 8 năm 2017). “An Engineered orco Mutation Produces Aberrant Social Behavior and Defective Neural Development in Ants”. Cell. 170 (4): 736–747.e9. doi:10.1016/j.cell.2017.06.051. PMC 5587193. PMID 28802043.
- ^ Trible W, Olivos-Cisneros L, McKenzie SK, Saragosti J, Chang NC, Matthews BJ, Oxley PR, Kronauer DJ (tháng 8 năm 2017). “orco Mutagenesis Causes Loss of Antennal Lobe Glomeruli and Impaired Social Behavior in Ants”. Cell. 170 (4): 727–735.e10. doi:10.1016/j.cell.2017.07.001. PMC 5556950. PMID 28802042.
- ^ Kistler KE, Vosshall LB, Matthews BJ (tháng 4 năm 2015). “Genome engineering with CRISPR-Cas9 in the mosquito Aedes aegypti”. Cell Reports. 11 (1): 51–60. doi:10.1016/j.celrep.2015.03.009. PMC 4394034. PMID 25818303.
- ^ Friedland AE, Tzur YB, Esvelt KM, Colaiácovo MP, Church GM, Calarco JA (tháng 8 năm 2013). “Heritable genome editing in C. elegans via a CRISPR-Cas9 system”. Nature Methods. 10 (8): 741–743. doi:10.1038/nmeth.2532. PMC 3822328. PMID 23817069.
- ^ Jiang W, Zhou H, Bi H, Fromm M, Yang B, Weeks DP (tháng 11 năm 2013). “Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice”. Nucleic Acids Research. 41 (20): e188. doi:10.1093/nar/gkt780. PMC 3814374. PMID 23999092.
- ^ Wang H, Yang H, Shivalila CS, Dawlaty MM, Cheng AW, Zhang F, Jaenisch R (tháng 5 năm 2013). “One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering”. Cell. 153 (4): 910–918. doi:10.1016/j.cell.2013.04.025. PMC 3969854. PMID 23643243.
- ^ Soni D, Wang DM, Regmi SC, Mittal M, Vogel SM, Schlüter D, Tiruppathi C (tháng 5 năm 2018). “Deubiquitinase function of A20 maintains and repairs endothelial barrier after lung vascular injury”. Cell Death Discovery. 4 (60): 60. doi:10.1038/s41420-018-0056-3. PMC 5955943. PMID 29796309.
- ^ Guo X, Li XJ (tháng 7 năm 2015). “Targeted genome editing in primate embryos”. Cell Research. 25 (7): 767–768. doi:10.1038/cr.2015.64. PMC 4493275. PMID 26032266.
- ^ Baltimore D, Berg P, Botchan M, Carroll D, Charo RA, Church G, Corn JE, Daley GQ, Doudna JA, Fenner M, Greely HT, Jinek M, Martin GS, Penhoet E, Puck J, Sternberg SH, Weissman JS, Yamamoto KR (tháng 4 năm 2015). “Biotechnology. A prudent path forward for genomic engineering and germline gene modification”. Science. 348 (6230): 36–38. Bibcode:2015Sci...348...36B. doi:10.1126/science.aab1028. PMC 4394183. PMID 25791083.
- ^ Larson MH, Gilbert LA, Wang X, Lim WA, Weissman JS, Qi LS (tháng 11 năm 2013). “CRISPR interference (CRISPRi) for sequence-specific control of gene expression”. Nature Protocols. 8 (11): 2180–2196. doi:10.1038/nprot.2013.132. PMC 3922765. PMID 24136345.
- ^ Yan MY, Yan HQ, Ren GX, Zhao JP, Guo XP, Sun YC (tháng 9 năm 2017). “CRISPR-Cas12a-Assisted Recombineering in Bacteria”. Applied and Environmental Microbiology. 83 (17). doi:10.1128/AEM.00947-17. PMC 5561284. PMID 28646112.
- ^ Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F (tháng 10 năm 2015). “Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system”. Cell. 163 (3): 759–771. doi:10.1016/j.cell.2015.09.038. PMC 4638220. PMID 26422227.
- ^ Fonfara I, Richter H, Bratovič M, Le Rhun A, Charpentier E (tháng 4 năm 2016). “The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA”. Nature. 532 (7600): 517–521. Bibcode:2016Natur.532..517F. doi:10.1038/nature17945. PMID 27096362. S2CID 2271552.
- ^ Kim H, Kim ST, Ryu J, Kang BC, Kim JS, and Kim SG (tháng 2 năm 2017). “CRISPR/Cpf1-mediated DNA-free plant genome editing”. Nature Communications. 8 (14406): 14406. Bibcode:2017NatCo...814406K. doi:10.1038/ncomms14406. PMC 5316869. PMID 28205546.
- ^ “Cpf1 Nuclease”. abmgood.com. Truy cập ngày 14 tháng 12 năm 2017.
- ^ Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DB, và đồng nghiệp (tháng 8 năm 2016). “C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector”. Science. 353 (6299): aaf5573. doi:10.1126/science.aaf5573. PMC 5127784. PMID 27256883.
- ^ Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, và đồng nghiệp (tháng 4 năm 2017). “Nucleic acid detection with CRISPR-Cas13a/C2c2”. Science. 356 (6336): 438–442. doi:10.1126/science.aam9321. PMC 5526198. PMID 28408723.
- ^ Gootenberg JS, Abudayyeh OO, Kellner MJ, Joung J, Collins JJ, Zhang F (tháng 4 năm 2018). “Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6”. Science. 360 (6387): 439–444. doi:10.1126/science.aaq0179. PMC 5961727. PMID 29449508.
- ^ Iwasaki RS, Batey RT (2020). “SPRINT: a Cas13a-based platform for detection of small molecules”. Nucleic Acids Research (bằng tiếng Anh). doi:10.1093/nar/gkaa673. PMID 32797156.
- ^ Ledford, Heidi (4 tháng 6 năm 2015). “CRISPR, the disruptor”. Nature. 522 (7554): 20–24. doi:10.1038/522020a. ISSN 1476-4687. PMID 26040877.
- ^ Snyder, Bill (ngày 21 tháng 8 năm 2014). “New technique accelerates genome editing process”. research news @ Vanderbilt. Nashville, Tennessee: Vanderbilt University.
- ^ Hendel A, và đồng nghiệp (tháng 9 năm 2015). “Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells”. Nature Biotechnology. 33 (9): 985–9. doi:10.1038/nbt.3290. PMC 4729442. PMID 26121415.
- ^ Ledford H (tháng 3 năm 2016). “CRISPR: gene editing is just the beginning”. Nature. 531 (7593): 156–9. doi:10.1038/531156a. PMID 26961639.
- ^ Maxmen, Amy (tháng 8 năm 2015). “The Genesis Engine”. WIRED. Truy cập ngày 5 tháng 6 năm 2016.
- ^ a b c d e f g h i j k l m n o Travis J (ngày 17 tháng 12 năm 2015). “Breakthrough of the Year: CRISPR makes the cut”. Science Magazine. American Association for the Advancement of Science.
- ^ Ledford H (tháng 6 năm 2015). “CRISPR, the disruptor”. Nature. 522 (7554): 20–4. doi:10.1038/522020a. PMID 26040877.
- ^ Hille F, Charpentier E (tháng 11 năm 2016). “CRISPR-Cas: biology, mechanisms and relevance”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 371 (1707): 20150496. doi:10.1098/rstb.2015.0496. PMC 5052741. PMID 27672148.
- ^ a b c Barrangou R, Marraffini LA (tháng 4 năm 2014). “CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity”. Molecular Cell. 54 (2): 234–244. doi:10.1016/j.molcel.2014.03.011. PMC 4025954. PMID 24766887.
- ^ Tyson GW, Banfield JF (tháng 1 năm 2008). “Rapidly evolving CRISPRs implicated in acquired resistance of microorganisms to viruses”. Environmental Microbiology. 10 (1): 200–207. doi:10.1111/j.1462-2920.2007.01444.x. PMID 17894817.
- ^ a b c d e f g h i j k l m n “Five big mysteries about CRISPR's origins”. Heidi Ledford. Nature. ngày 12 tháng 1 năm 2017. Truy cập ngày 8 tháng 6 năm 2018.
- ^ a b “CRISPR/Cas9 – molecular scissors made of enzyme and RNA”. Hiệp hội Max Planck. Truy cập ngày 8 tháng 6 năm 2018.
- ^ a b c d “Palindromes in the genome”. Max Planck Society. Truy cập ngày 8 tháng 6 năm 2018.
- ^ a b c d e f “Natural functions of CRISPR-Cas”. Max Planck Society. Truy cập ngày 8 tháng 6 năm 2018.
- ^ a b c d “Functioning of CRISPR-Cas9”. Max Planck Society. Truy cập ngày 8 tháng 6 năm 2018.
Liên kết ngoài
[sửa | sửa mã nguồn]| Wikimedia Commons có thêm hình ảnh và phương tiện truyền tải về CRISPR. |
| Scholia có topic thông tin về CRISPR. |
- Advanced Gene Editing: CRISPR-Cas9 Congressional Research Service
- Jennifer Doudna talk: Genome Engineering with CRISPR-Cas9: Birth of a Breakthrough Technology
- Tổng quan về mọi thông tin cấu trúc có sẵn trên PDB cho UniProt: Q46901 (CRISPR system Cascade subunit CasA) tại PDBe-KB.
- Tổng quan về mọi thông tin cấu trúc có sẵn trên PDB cho UniProt: P76632 (CRISPR system Cascade subunit CasB) tại PDBe-KB.
- Tổng quan về mọi thông tin cấu trúc có sẵn trên PDB cho UniProt: Q46899 (CRISPR system Cascade subunit CasC) tại PDBe-KB.
- Tổng quan về mọi thông tin cấu trúc có sẵn trên PDB cho UniProt: Q46898 (CRISPR system Cascade subunit CasD) tại PDBe-KB.
- Tổng quan về mọi thông tin cấu trúc có sẵn trên PDB cho UniProt: Q46897 (CRISPR system Cascade subunit CasE) tại PDBe-KB.
